
|
||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
||||||||||||||||
|
Location:Home > 実験手法別製品・技術情報 > セルラーサイエンス |
||||||||||||||||
IN Cell Analyzer 1000を用いた神経幹細胞分化の定量
|
||||||||||||||||
| Image | Human count | Standard deviation | Developer Toolbox count | |
|---|---|---|---|---|
| DAPI | 1 2 3 |
68.83 19.17 35.33 |
7.52 0.75 4.93 |
73 18 36 |
| MAP2 | 1 2 3 |
18.00 12.33 12.83 |
2.61 1.03 3.06 |
18 12 10 |
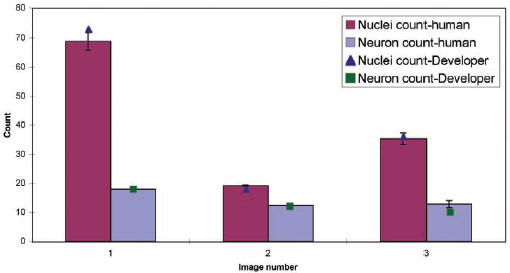
図1. マニュアルカウントとDeveloper Toolboxによるカウント結果の比較。
棒グラフはマニュアルカウント、ポイント(▲、■)はDeveloper Toolboxによるカウント結果です。
棒グラフ上部分にあるバーは、95%を信頼区間とした場合のエラーを示しています(n=6)
Developer Toolboxによる解析
それぞれのイメージをDeveloper Toolboxにより認識しました(図2)。認識前のイメージは左側、認識後のイメージは右側です。赤い領域は測定対象を示し、緑の領域は測定対象外を示しています。核のイメージ(Nuclear)はDAPIによる染色です。神経細胞(Neuronal)のイメージは一次抗体に抗MAP2抗体を、二次抗体にanti-rabbit Cy5 conjugated antibodyを使用して取得しました。
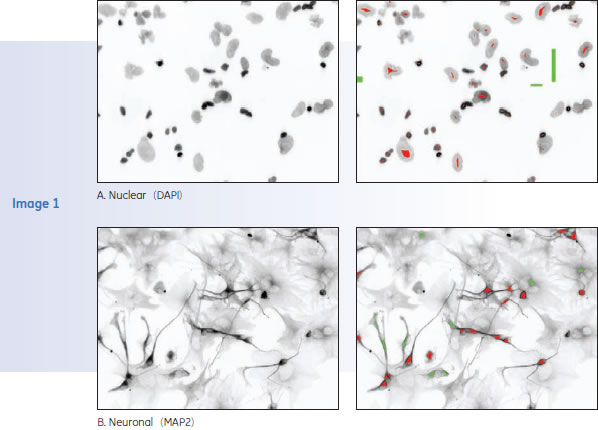


図2. Developer Toolboxによる画像認識の様子
3つの画像領域(Image1, 2, 3)を対象とした認識結果です。同領域について、DAPI 染色による核の認識結果(A, C, E)と、抗MAP2抗体による分化した神経経細の認識結果(B, D, F)を示しました。
IN Cell Analyzer 1000で取得したシグナル(各左側の画像)から、Developer Toolboxにより核、神経細胞を認識しました(各右側の画像)。
細胞形態が複雑な箇所や細胞が密集している箇所であっても、神経細胞を正確に認識していることがわまります。
※Developer Toolboxで神経細胞を認識する流れについては、「Developer Toolbox による画像認識」をご参照ください。
神経細胞の定量例
10 µMレチノイン酸の処理時間を変えてNTERA-2細胞を培養し、神経細胞分化の定量を行いました。測定するタイムポイントにおいて、分裂阻害剤を添加した培地で6日間培養した後、固定、染色を行い、解析に用いました。
全細胞における神経細胞の割合を示したものが図3、全細胞と神経細胞をカウントした結果が図4です。
図3では、レチノイン酸処理が17日間を超えると、分化した神経細胞の割合が減少していることが示されています。これは、レチノイン酸による増殖抑制(神経細胞への分化促進)を受けていない細胞の増殖によるものであることが図4からわかります。
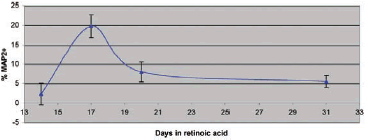
図3. 分化した神経細胞の割合
それぞれのタイムポイントで6ウェルを撮影し、1ウェルにつき5枚の画像を取得し数値化しました。エラーバーは標準偏差を示します。
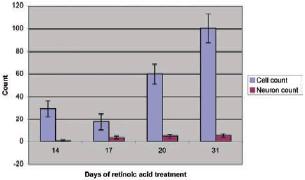
図4. 細胞全体の数と分化した神経細胞の数
神経細胞への分化はレチノイン酸添加後、17日目から起こりました。(エラーバーは標準偏差)
Developer Toolbox による画像認識
画像から有意義なデータを得るには、極度に強度の高いシグナルや対象物が重なり合って検出されるような画像からでも、興味のある対象領域とその他の領域を正確に認識し分類できることが重要です。
対象を分類するための最良の方法は、ひとつひとつの要素(例.大きさや輝度)ごとに対象を選び出し、それらを組み合せる(マクロを使用する)ことです。対象物を分類することができたら、次に対象物の特性を表すデータ(シグナル強度、長さ、形態など)を計測します。
画像を目で観察し、目的の細胞群(本報では神経細胞)とそれ以外の集団とを見分けるための変化を見いだします。2つの集団間で常に異なっている変化を見つけ、集団を区別するための判断基準とします。
以下に、Developer Toolboxによる、分化した神経細胞のカウント解析の流れを示します。一度解析アルゴリズムを構築してしまえば、自動で解析が可能になります。
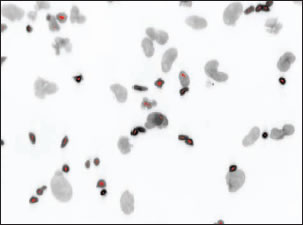
Step 1. 小さな核を選択的に認識します。
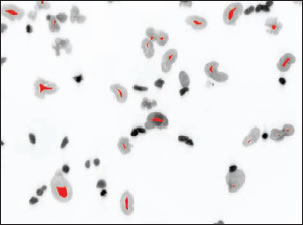
Step 2. 大きな核を選択的に認識します。
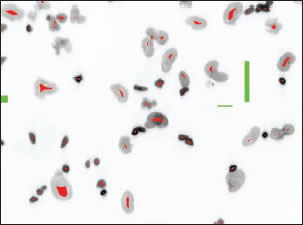
Step 3. 小さな核と大きな核の画像を組み合わせすべての核を認識します。
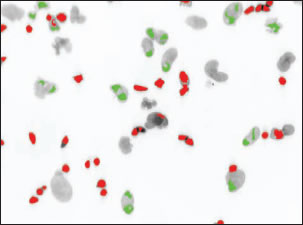
Step 4. 神経細胞の核は小さいため、面積の大きい核(図中、緑)を除外し、面積の小さい核のみを認識(図中、赤)するようにします。

Step 5. 検出したMAP2-positive細胞のシグナルの中から、Step 4で認識した核と重なりあうシグナルのみを分離し、神経細胞としてカウントします(図中、赤)。このステップにより、ひとつの核につきひとつの神経細胞を対応づけることができます。
Step 4で核認識されていないシグナル(図中、緑)は、神経細胞としてはカウントされません。
Conclusion
幹細胞、細胞分化、前駆細胞や分化細胞由来の幹細胞などを対象とした研究において、影響を与える化合物や生体分子の効率的なスクリーニングには、解析の自動化が求められます。
本報では、異なるタイプの細胞が混在している画像をもとにして、Developer Toolboxを用いた画像解析により対象となる細胞の定量解析を行った例をご紹介しました。いくつかの分離ファクターを組み合せた解析アルゴリズムを構築することで、高密度な細胞集団からでも、神経幹細胞の神経細胞への分化の度合いを正確に自動計測することが可能になりました。このような細胞画像をもとにした自動解析によるアプローチは、幹細胞や発生生物学に関する他の一般的なアプリケーションにも適用できます。
お問合せフォーム
※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。
お問い合わせありがとうございます。
後ほど担当者よりご連絡させていただきます。
© 2024 Cytiva
