|
|||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|||||||||||||||||||||||||||||||||||
|
Location:Home > サポート・保守情報 > お客さまの声・データ紹介 |
|||||||||||||||||||||||||||||||||||
お客さまの声・データ紹介 - GenomiPhi™培養困難な海水中単細胞真核生物のPCRフォロー理由推奨プロトコールより少ない100 細胞以下での増幅も含まれているため フォロー状況可能であれば増幅は1000 細胞以上で行うことをおすすめした上で、100細胞以下のサンプルから増幅したGenomiPhi™増幅産物を使用したPCR産物が、本当に目的のものかを、シークエンシング等で確認することをおすすめしています。 出発材料天然の海水サンプル中の単細胞真核生物Ebria tripartita の18S rDNA配列 GenomiPhi™増幅産物を使用したアプリケーション名PCR 実験方法1. GenomiPhi™使用ステップ(A)使用機器・試薬
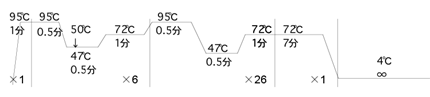
(B)プロトコール 海水サンプル中からEbria tripartita の細胞をマイクロピペットで単離し、1、10、20、50、100細胞を用いて次のような組成で最初の変性反応(95 ℃、3分)をおこなった。
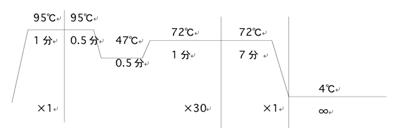
上記反応を行ったチューブにあらかじめ氷上で調製した酵素反応液(Reaction buffer 9 µl, Enzyme mix 1 µl)を加え、30 ℃で26時間反応させた。 2. その後のアプリケーションのステップ(A)使用機器・試薬
(B)プロトコール
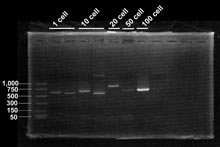
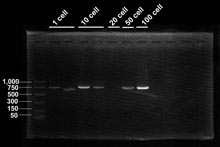
反応
2% TAEゲル泳動 PCR marker (Promega社)および2nd PCR産物それぞれ20 µlを2% TAEゲル中で泳動し、エチジウムブロマイドにて染色を行った。 結果 (下記画像をクリックすると拡大表示されます)結果に対するコメントEbria tripartita は培養が困難であるため、これまで遺伝子の配列がわからず、分類学的に所属位置が不明の生物である。以前、一細胞を用いてPCRに挑戦していたが、PCR反応による増幅産物が得られなかった経験があるので、今回のGenomiPhi™の適用を試みた。 お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
|||||||||||||||||||||||||||||||||||
© 2026 Cytiva