|
||
|---|---|---|
|
|
||
|
Location:Home > 実験手法別製品・技術情報 > セルラーサイエンス |
||
IN Cell Analyzer による遺伝毒性試験コメットアッセイの実施はじめに薬剤、化学物質や電離放射線により生じるDNA 損傷を評価する遺伝毒性試験は医薬品開発の重要な段階であり、現在さまざまな方法が使用されています(Walmsley, 2005)。また疫学研究、環境モニタリングにおいても広く使用されています(Møller et. al, 2000)。 中でもコメットアッセイと一般的に呼ばれる単一細胞電気泳動法による評価法(Ostling and Johanson, 1984)はin vitro 小核試験などの遺伝毒性試験と異なり分裂期細胞を必要とせず、すべての細胞のDNA 損傷を測定できます。細胞数や細胞の種類の制約により他のDNA 損傷の測定法を使用できない場合にも本アッセイ法を使用できます。特に高度に分化した非分裂細胞に有効です。薬剤の安全性試験から環境モニタリングまで広範囲のアプリケーションで適用可能です。 この手法では、細胞をアガロースゲルに封入、溶解し、DNAを変性させた後に電気泳動を行い、損傷したDNA を検出します。続いて細胞をDNA 結合蛍光色素で染色して、顕微鏡画像を取得します。DNA 損傷のない細胞ではDNA が細胞核に存在したままであり、顕微鏡画像で明るいスポットとして現れます。DNA 損傷が生じた細胞では、DNA 鎖の切断が起こり、損傷DNA の溶解と変性によりDNA 構造内のスーパーコイルがほどけて、DNA が核から電気泳動液中に遊離します。その後染色し、顕微鏡で観察すると、遊離したDNA は核の頭から流れ出す尻尾(テイル)のようにみえます。彗星(コメット)のようにみえることからアッセイの名前になりました。 本アプリケーションノートではIN Cell Analyzer 1000 で取得したコメットアッセイ画像を、画像解析パッケージであるIN Cell Investigator Developer Toolbox にて解析しました。Developer Toolbox は、個々の実験に適した分析プロトコールを柔軟に設定でき、幅広いアプリケーションに対応します。
実験方法培養したJurkat 細胞(白血病T 細胞株)を回収して、2×105 cells/ml になるようにRPMI 1640 培地で希釈しました。細胞をPBS で2 回洗浄し、H2O2 100 μM 含有または非含有のPBS に再懸濁して、遮光下4℃で20 分間インキュベートしました。処理細胞を37℃の低融点アガロース(LMA; Trevigen)で10 倍に希釈し、ピペッティングにより穏やかに混和しました。75 μl のLMA 中の細胞を37℃に保温したコメットアッセイ用スライド(Trevigen)に滴下し、スライドを4℃で10 分間放置してLMA を固化しました。スライドを遮光下4℃で60 分間細胞溶解液(Trevigen)に浸しました。溶解後にスライドを拭き、遮光下室温で60 分間アルカリ溶液(0.3 M NaOH、1 m MEDTA)に浸しました。次にスライドを拭いて、1×TBE 電気泳動緩衝液を用いて室温で5 分間、2 回洗浄しました。電気泳動では、スライドを水平電気泳動槽中で1×TBE に浸し、1 ボルト/cmで10 分間通電しました。スライドを70%エタノール中に5 分間放置してDNA を固定し、室温で一晩乾燥させました。画像解析用に、SYBR GREEN(Trevigen)を4℃で5分間処理してDNA を染色し、余分な染色液を吸い取りました。乾燥したスライドを用いて、4×対物レンズ、480 ~ 40 nm 励起フィルター、535 ~ 50 nm 蛍光フィルターでIN Cell Analyzer1000 により画像を取得しました。
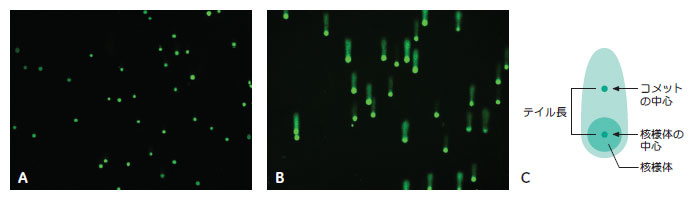
結果ヒドロキシラジカル生成を介してDNA 損傷を起こすことが知られている過酸化水素(H2O2)で処理し、IN Cell Analyzer 1000 を使ってコメットアッセイスライドの画像(図1)を取得しました。得られた画像はIN Cell Investigator Developer Toolbox を使って解析しました。
まず、核様体部分の核に残ったDNA と移動したDNA のコメット部分とを区別して認識しました。次に核様体部分とコメット部分の蛍光強度を測定し、それぞれの蛍光強度の中心位置を決定しました(図1 C)。 テイル内の%DNA:核様体DNA(核様体のみの蛍光)とコメットDNA(全蛍光)の蛍光強度を用いて、コメットテイル内のDNA 染色強度の割合を以下の式で算出しました。 %DNA = [ コメット染色強度-核様体染色強度]/コメット染色強度×100 テイル長:結合させた核様体とコメットの中心位置(各画像の左下隅からの距離をx, y 座標としてμmで表示)を用いて対の対象間の距離を算出しました(図1 C)。 
テイルモーメント:テイル内の%DNA とテイル長を組合せてDNA 損傷の尺度を求めました。 テイルモーメント = [ テイル内の%DNA]×[ テイル長]
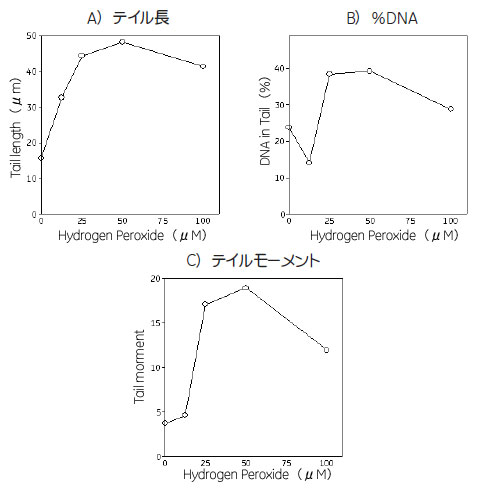
各濃度のH2O2 に曝露した細胞から取得した画像の解析により、明らかな用量依存性のDNA損傷の増強が示されました(図3)。
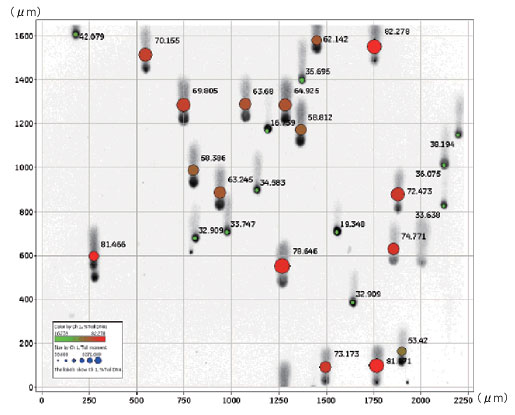
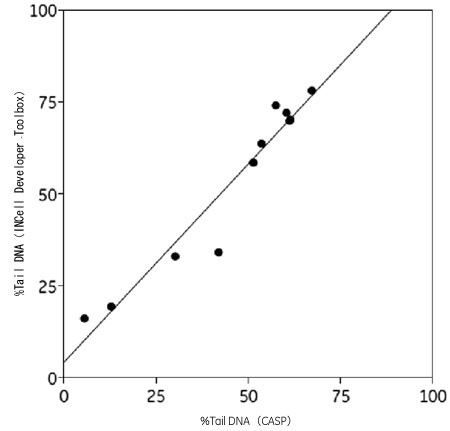
H2O2で処理した細胞の画像を用いて、Developer Toolbox で解析した結果とCASP(各コメットを手動で選択する標準的なコメットアッセイ解析パッケージ:http://casp.sourceforge.net)を用いて解析した結果を比較しました。 コメットテイルDNA の蛍光強度データの比較で、DeveloperToolbox を用いた完全自動解析とCASPソフトウェアを用いた従来の手動解析との間で良好な相関関係が示されました(図5)。
以上より、遺伝毒性評価の小核試験の代替法としてIN Cell Analyzer 1000 を用いたコメットアッセイを使用できることが分かりました。さらに、この手法では他のマーカーを追加することが可能です。例えばBrdU(McGlynn et.al., 1999)染色を追加すれば新たに複製されたDNA 中のDNA 損傷を測定でき、蛍光in situ ハイブリダイゼーション(Rapp et.al., 2005)と組合わせれば、特定の遺伝子へのDNA 損傷を測定できる可能性があります。
結論
お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
||
© 2026 Cytiva