|
||
|---|---|---|
|
|
||
|
Location:Home > 実験手法別製品・技術情報 > ゲノムDNAの精製 |
||
FTAカード上に保存された血液、培養細胞、動物、植物の組織サンプルからのRNAspin MiniによるRNA精製(4)植物研究 序論植物からの核酸抽出は、細胞壁の物理的な破壊や、さまざまな試薬を必要とします。必要となる試薬の多くは、核酸に与えるダメージが大きいため、下流のアプリケーションの妨げになることがあります。通常は、動物細胞からRNAを精製するように、液体窒素中で組織をすり潰してして破砕します。RNAを保存するには、サンプル採取後、直ちに液体窒素で凍結させる必要があり、サンプルの採取場所が戸外や実験室外である場合のボトルネックとなります。 本項では、全く異なる2種類の植物遺伝子の発現解析でのFTAカードの有用性をご紹介します。 1つ目は、シロイヌナズナのプロテインホスファターゼPP2A C4サブユニット(At3g58500)をノックアウトした変異体SALK_035009です。 2つは、シロイヌナズナDR5::GUSプロモーターレポーター導入(6)変異体です。GUS(βグルクロニダーゼ)は特定の条件で植物組織の色を青色に変化させるレポーター遺伝子のひとつです。本項では、オーキシンでGUSレポーター遺伝子の発現を誘導します。
材料および方法サンプル採取葉(PP2A植物体)または幼苗(DR5::GUSプロモーターリポーター導入シロイヌナズナの幼苗で、一般的なオーキシンである1-ナフタレン酢酸でGUSレポーター遺伝子の発現を誘導したものと、GUSレポーター遺伝子の発現を誘導していないものを使用)をFTAカード上に置きパラフィルムで覆いました。小さなハンマーでその上から軽く組織をたたきつぶし*、液体をカードの裏までしみ込ませました。残った植物組織をカードから取り除いた後、室温で3時間乾燥させました。サンプルアプライから5日以内にRNAを精製しました。 * 動物細胞とプロトコールが異なる箇所です。 RNA精製とDNase処理illustra™ RNAspinを用いて、前述した方法で、FTAカード上に採取した植物の葉や幼苗からトータルRNAを単離しました。Harris Uni-Core™ディスポーザブルパンチ(5.0 mm)を用いて、各乾燥済みサンプルスポットの中央からディスクを打ち抜きました。ライセートの採取には、18ゲージのニードルを使用しました。製造者標準プロトコールに従いましたが、カラム上でのDNase処理は省略しました*。抽出後、RNAを直ちにDNA-freeキット(Ambion社)を用い、メーカーの取扱説明書にしたがってDNase処理を行い、抽出したDNAを完全に切断しました。 * 動物細胞とプロトコールが異なる箇所です。ご注意ください。 発現解析7900HT Real Time PCRシステム(ライフテクノロジーズ社)およびTaqMan™ RNA to Ctキット(ライフテクノロジーズ社)を用いてRT-qPCR によりRNAの定量を行いました。RNA中にゲノムDNAの混入がないことを、ライフテクノロジーズTIP41様遺伝子に特異的なプライマー(後述)を使用しPower SYBR Green Master Mix(ライフテクノロジーズ社)で確認しました。 Primer Expressソフトウェア(ライフテクノロジーズ社)を使用して以下の遺伝子に特異的なプライマーを設計し、Integrated DNA Technologies社から購入しました。
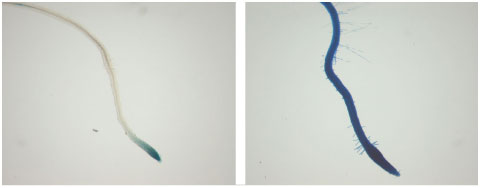
RT-qPCRによって得たCq値を用いて、内部標準遺伝子(TIP41様遺伝子)に対する標的遺伝子(PP2AまたはGUS)の発現をΔΔCq法により計算しました(8)。 免疫組織化学的染色発現解析のためDR5::GUSプロモーターレポーター導入シロイヌナズナの幼苗を採取し、コントロールとGUS遺伝子発現誘導済み幼苗の各10サンプル以上をMartin et al.(9)のプロトコールに従い染色しました。バキュームを使用して、X-Gluc基質を幼苗に浸潤させました。さらに、非真空下にて37℃で4時間、基質溶液中で幼苗をインキュベートしてから顕微鏡で観察しました。
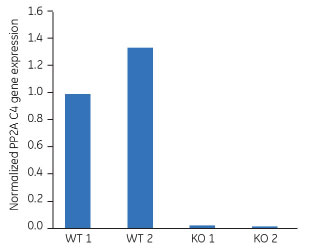
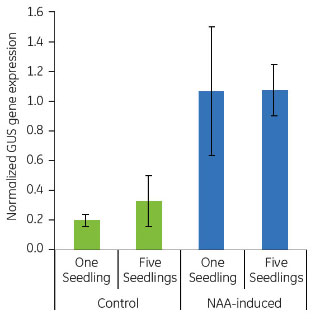
結果と考察ノックアウト植物のPP2A C4発現解析すべてのRNAサンプルで、TIP41様遺伝子とPP2A C4遺伝子の転写産物をRT-qPCRにより増幅しました。使用したTotal RNA量がサンプル間で異なるので、これを補正するためPP2A C4の発現量をTIP41様遺伝子の発現量でノーマライズしました。図3は、PP2A C4遺伝子でのノーマライズ後の、野生株(WT)とノックアウト株(KO)の2サンプルの発現量を示したものです。 幼苗のGUS発現解析すべてのRNAサンプルで、TIP41様遺伝子とGUS遺伝子の転写産物をRT-qPCRにより増幅しました。前述した方法によって、GUS遺伝子の発現量は、TIP41様遺伝子の発現量でノーマライズしました。DR5::GUSコントロールと1-ナフタレン酢酸(NAA)発現誘導済みシロイヌナズナにおけるGUS遺伝子の発現量を図4に示します。 図4は、1個体の幼苗からRT-qPCRに十分なRNAが得られることを示しています。植物1個体の相対的遺伝子発現量と植物5個体プールの相対的遺伝子発現量の比較から、使用した組織の量(使用した幼苗個体数)は、相対的遺伝子発現量に大きく影響しないことがわかりました(図4)。 1-ナフタレン酢酸(NAA)誘導DR5::GUS幼苗で観察されたGUS遺伝子発現量が、DR5::GUSコントロールと比較して高いことは、以前の研究(6)によって示されており、免疫組織染色でも確認ができました(図5)。
植物研究:結論FTAカードで保存した植物組織由来のRNAは、発現解析に使用できます。FTAカード上で保存した葉と幼苗から、illustra™ RNAspin MiniでRNAを精製し、発現解析したところ、野生株とノックアウト変異体の間で特定のmRNA量に明らかな差が見られました。また、レポーター遺伝子構築物を使用した実験からは、植物を化学誘導物質に暴露することで特定のmRNA量が高くなることがわかりました。
お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
||
© 2026 Cytiva