|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Location:Home > サポート・保守情報 > お客さまの声・データ紹介 |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
お客さまレビュー/データ紹介 illustra™ GenomiPhi™ DNA Amplification Kit その3ゲノムDNA 増幅試薬
illustra™ GenomiPhi™ DNA Amplification Kit は、illustra™ TempliPhi™ DNA Amplification Kit と同様に高い校正能をもつPhi 29酵素を用いており、高精度にゲノムDNAを複製することができます。2007年に改良型が販売されており、目的別に2種類のキットをお選びいただけます。
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 添付の10 x buffer | 2 µl |
| dNTP (2.5 mM each) | 1.6 µl |
| primer-1 (10 µM) | 0.3 µl |
| primer-2 (10 µM) | 0.3 µl |
| TaKaRa Taq (5 U/µl) | 0.1 µl |
| genome DNA (約1 ng/µl) またはGenomiPhi™反応液 |
1 µl |
| 滅菌水 | 14.3 µl |
| 計 | 20 µl |
以下のPCR反応を行った。
| 95℃ | 1.5 min | ||
| 95℃ | 30 s | \ | |
| 63℃ | 60 s | 25 サイクル | |
| 72℃ | 30 s | / | |
| 72℃ | 2 min | ||
| 4℃ | over night |
【使用機器・試薬】
- 機器:Eppendorf™ Mastercycler gradient
- 試薬:宝酒造 TaKaRa Taq
※PCR反応後、5 µl をアガロース泳動(2% agarose in 1 x TBE)に供した。
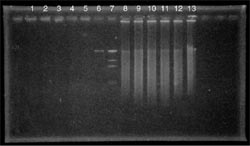
結果 (画像をクリックすると拡大表示されます)
| lane 1: | 卵 No.1 | ゲノムDNA | 1 µl (1.3 ng) |
|---|---|---|---|
| lane 2: | 卵 No.2 | ゲノムDNA | 1 µl (1.2 ng) |
| lane 3: | 卵 No.3 | ゲノムDNA | 1 µl (0.9 ng) |
| lane 4: | 卵 No.4 | ゲノムDNA | 1 µl (1.6 ng) |
| lane 5: | 卵 No.5 | ゲノムDNA | 1 µl (1.3 ng) |
| lane 6: | control DNA | 1 µl (10 ng) | |
| lane 7: | マーカー | ||
| lane 8: | 卵 No.1 | GenomiPhi™反応液 | 1 µl |
| lane 9: | 卵 No.2 | GenomiPhi™反応液 | 1 µl |
| lane 10: | 卵 No.3 | GenomiPhi™反応液 | 1 µl |
| lane 11: | 卵 No.4 | GenomiPhi™反応液 | 1 µl |
| lane 12: | 卵 No.5 | GenomiPhi™反応液 | 1 µl |
| lane 13: | control DNA | GenomiPhi™反応液 | 1 µl |
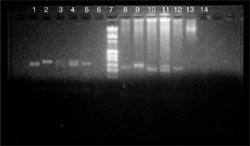
| lane 1: | 卵 No.1 | ゲノムDNA | 1 µl (1.3 ng) | => PCR反応 |
|---|---|---|---|---|
| lane 2: | 卵 No.2 | ゲノムDNA | 1 µl (1.2 ng) | => PCR反応 |
| lane 3: | 卵 No.3 | ゲノムDNA | 1 µl (0.9 ng) | => PCR反応 |
| lane 4: | 卵 No.4 | ゲノムDNA | 1 µl (1.6 ng) | => PCR反応 |
| lane 5: | 卵 No.5 | ゲノムDNA | 1 µl (1.3 ng) | => PCR反応 |
| lane 6: | control DNA | 1 µl (10 ng) | => PCR反応 | |
| lane 7: | マーカー | |||
| lane 8: | 卵 No.1 | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 9: | 卵 No.2 | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 10: | 卵 No.3 | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 11: | 卵 No.4 | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 12: | 卵 No.5 | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 13: | control DNA | GenomiPhi™反応液 | 1 µl | => PCR反応 |
| lane 14: | template なし | (negative control) |
結果に対するコメント
PCR反応のtemplateとして、ゲノムDNA溶液1 µl をそのまま使った場合(lane 1-5)と、ゲノムDNA溶液1 µl をGenomiPhi™ DNA Amplification Kit 反応に供し、その反応液20 µl のうちの1 µl を使った場合(lane 8-12)とで、ほぼ同程度の量のPCR産物(マイクロサテライトを含む領域)が得られた。
ゲノムDNA溶液はtotal 100 µl あるので、GenomiPhi™ DNA Amplification Kit を使用しない場合は100回のPCRが行えるのに対して、GenomiPhi™ DNA Amplification Kit を使用した場合は2000回ものPCR反応が行えることになる。従って、調べたいマイクロサテライトのlocusが非常に多い場合には、GenomiPhi™ DNA Amplification Kit は極めて有効な手段であると考える。
農業・特定生物産業技術研究機構 九州沖縄農業研究センター 作物機能開発部 育種工学研究室
土門 英司 先生
GenomiPhi™ DNA Amplification Kit の良いところ
- PCRと比較してslippageの頻度が低いと考えられる
- DNA抽出が困難な植物についても、抽出された微量のDNAを増幅することができる
目的
Simple Sequence Repeat (SSR) 多型検出
サンプル
微量のイグサ(Juncus effusus L.)ゲノムDNA
GenomiPhi™ DNA Amplification Kit を用いた実験方法
- イグサ(Juncus effusus L.)、4系統のゲノムDNA各5ngを供試し、標準的なプロトコールに従ってABI GeneAmp™ PCR System 9700を使用して、30度18時間の増幅反応を行った。
- 増幅産物20 µl をTE Buffer(10 mM Tris-Cl、1mM EDTA)で2倍希釈し、Hoechist33258染色液を使用して蛍光分光光度計(日立、F-4010)でDNA濃度を測定した。
【使用機器・試薬】
- ABI GeneAmp™ PCR System 9700
- Hoechist 33258染色液
- 蛍光分光光度計(日立、F-4010)
GenomiPhi™ DNA Amplification Kit によるDNA増幅後のステップ
- SSR 多型解析
- 増幅産物60 ngとCTAB法で抽出したゲノムDNAを供試して、SSR多型解析を行った。SSR多型解析には4種類のプライマーセットを使用した。
- PCR産物は3%アガロースゲルで電気泳動しEthidium bromideで染色した。
【使用機器・試薬】
- ABI GeneAmp™ PCR System9700
- アガロースゲル電気泳動装置
結果
(1) MDAにより全系統について約8 µg の増幅産物が得られた(下表)。1回のPCRに使用するゲノムDNAの所要量は30-60 ngであることから、130-270回ものPCRが可能である。増幅産物の濃度標準誤差も十分に小さく、安定な増幅が得られている。
| 品種 | 反復 | 濃度(ng/µl) | 標準誤差 | 収量(µg) |
|---|---|---|---|---|
| A | 3 | 194.5 | 2.83 | 7.8 |
| B | 3 | 186.3 | 0.84 | 7.5 |
| C | 3 | 209.7 | 10.29 | 8.4 |
| D | 3 | 219.1 | 1.14 | 8.8 |
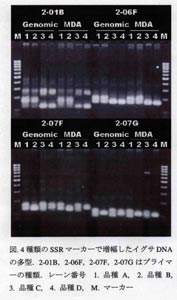
(2) SSR多型検出用のプライマーセットを使用してPCRを行った。結果を図に示す(下記画像をクリックすると拡大表示されます)
図に示したように、ゲノムDNAとMDAで増幅したDNAではSSRのパターンは品種間でほぼ保存されていた。一部、SSRマーカー2-07品種D(レーン4)についてはゲノムDNAを鋳型とした場合2本のバンドが増幅されるが、MDA由来のPCR産物ではそのうちの1本のバンドが薄くなる傾向が見られた。原因は不明である。
SSRを含む領域をゲノムDNAから増幅する場合、PCRのサイクルが多いと、アガロースゲル上のバンドが不鮮明になりがちである。これはDNA polymeraseのslippageによって反復部分でDNA断片の長さに違いが出ることが原因であると考えられる。本実験では、MDAで鋳型DNAを一旦増幅しているにもかかわらず、アガロースゲル上でのPCR産物のバンドの鮮明さはゲノムDNAから直接PCRを行った場合とほぼ同等である。
結果に対するコメント
- phi29DNA polymeraseによる等温増幅ではPCRと比較してslippageの頻度が低いものと考えられる。
- DNA抽出が非常に困難な植物についても、抽出された微量のDNAを増幅することができる。
北里大学 北里生命科学研究所 創薬科学部門 ゲノム情報学研究室
山下 敦士 先生、服部 正平 先生
GenomiPhi™ DNA Amplification Kit の良いところ
- 手間がかからない
- 今まで難培養で解析不可能であった生物種への応用
研究の背景
自然界には数十万種類もしくは百万種類以上の微生物が存在するといわれていますが、全ては同定されていません。さらに、同定された微生物についてもそれぞれ培養方法が確立されていないためにサンプルを集めることが難しく、生物学的な解析はごく一部の微生物に限られています。微生物は有用物質を生産する一方で、人や家畜などの動物へ感染して病気をもたらすなど私たちと深いかかわりを持っていますが、私たちが微生物について知っていることはまだほんのわずかです。
もし培養方法が確立されていない微生物の微量ゲノムDNAを試験管内にてGenomiPhi™ により増幅し、その全ゲノム情報を解読できれば、今まで未知であった新規な有用遺伝子を多数発見する糸口になります。その結果、病原細菌や抗生物質生産菌の解析は病原遺伝子や二次代謝産物に関連した遺伝子の解明など医療分野での貢献が期待できます。さらには、発酵細菌、海洋細菌、土壌細菌などのゲノム解析は有益でさまざまな遺伝子資源を提供でき、食生活(健康)、環境、エネルギーなどの人間社会に密着した分野への応用に新たな道を開くことが期待されます。
使用機器・試薬
- GenomiPhi™ 反応装置:インキュベーター
- シークエンサー:MegaBACE 4000
- シークエンシング反応キット:DYEnamic ET Terminator Cycle Sequencing kit for MegaBACE
GenomiPhi™ DNA Amplification Kit を用いた実験方法
まずはじめに、モデル実験系を構築するために、既知微生物である1.3 MbのMycoplasma penetrans より精製したゲノムDNAの希釈系列(10 ng~10 fg)を作り、それぞれGenomiPhi™ DNA Amplification Kit を用いて増幅したDNAからゲノムライブラリーを作製して、ショットガン・シークエンシングを行いました。
1 ng~1 pgまではほぼ同じ量のDNAがGenomiPhi™ DNA Amplification Kit によって増幅されていることが分かります(図1A)。これらからそれぞれせん断処理によってDNAを約2 kbpの断片に短くしてベクターへ組み込み(図1B)、ランダムに約1,000クローンを選んで両方向からシークエンシングしました。
結果
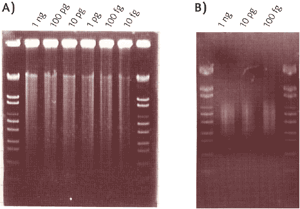
図1. M. penetrans ゲノムDNA希釈系列のGenomiPhi™による増幅
A)1 ng~10 fgの範囲で10倍希釈したゲノムDNA(国立感染症研究所・佐々木裕子先生よりご提供)をプロトコールにしたがってGenomiPhi™ DNA Amplification Kit による増幅を行い、アガロースゲル電気泳動により確認しました。1 ng~1 pgまではほぼ同様に増幅していますが、100 fg以下では増幅効率が低下しています。
B)
GenomiPhi™ DNA Amplification Kit 増幅DNAをせん断処理により約2 kbの断片にして、ベクターにライゲーションし、それぞれ約1,000クローンをショットガン・シークエンシングにより解析しました。
その結果、10 ngおよび 1 ngのゲノムDNAからGenomiPhi™ DNA Amplification Kit 増幅した場合、全てのクローンはM. penetrans 由来のもので、1 pgのゲノムDNA(~103個の微生物に相当)から増幅した場合でも約90%はM. penetrans 由来のクローンでした(表1、図2)。
| ゲノムDNA量 | 10 ng | 1 ng | 100 pg | 10 pg | 1 pg | 100 fg | 10 fg |
|---|---|---|---|---|---|---|---|
| 理論上の細胞数 | 6,713,522 | 671,352 | 67,135 | 6,714 | 671 | 67 | 7 |
| 解析したコロニー数 | 1,056 | 1,056 | 1,056 | 1,056 | 1,056 | 1,056 | 1,056 |
| シークエンス数(両鎖) | 2,112 | 2,112 | 2,112 | 2,112 | 2,112 | 2,112 | 2,112 |
| 成功リード数 | 1,727 | 1,519 | 1,275 | 1,408 | 1,239 | 1,244 | 1,132 |
| M. penetrans 由来のリード数 | 1,727 | 1,519 | 1,251 | 1,401 | 1,094 | 187 | 4 |
| 上記以外のリード数 | 0 | 0 | 24 | 7 | 145 | 1,057 | 1,128 |
1 pg以上のゲノムDNAを用いた場合、90%以上がM. penetrans 由来のクローンでした。
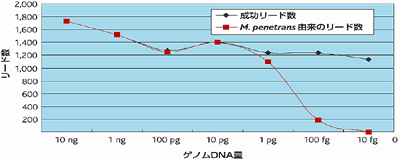
図2. ショットガン・シークエンシング解析のまとめ
表1をグラフ化した図です。数百分子相当のゲノムDNAからでもシークエンシング解析が可能であることがわかります。
残りの約10%のクローンはGenomiPhi™ DNA Amplification Kit の製造工程で用いる組換え大腸菌由来のDNAなどが含まれていました。また、これらのクローンがゲノムのどの領域をカバーしているのか確認したところ、ほぼ全域から片寄りなくシークエンシングができていることがわかりました。これはゲノムのほぼ全領域からDNAがランダムに増幅されていることを示唆しています(図3)。
図3. シークエンシング解析したクローンがカバーするM. penetransゲノム領域
いずれのクローン群でもほぼ全域に渡ってクローンを得ていることから、GenomiPhi™ DNA Amplification Kit はほぼ全域にわたってゲノムDNAを増幅していることが分かります。
以上のことから、大量のサンプルを集めなくてもGenomiPhi™ DNA Amplification Kit を用いてゲノムDNAを増幅すれば、今までは難培養で解析不可能であった微生物のゲノム解析を容易に進めることができるといえます。
GenomiPhi™ DNA Amplification Kit はDNA多型解析を容易にするだけでなく、ゲノムシークエンシング解析を進める上でも有用で、これまでに誰もが成し得なかった解析を大きく推進し、ひいては生物学のニューパラダイム創出の引き金になることが期待されます。
お問合せフォーム
※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。
お問い合わせありがとうございます。
後ほど担当者よりご連絡させていただきます。
© 2026 Cytiva