|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Location:Home > 実験手法別製品・技術情報 > BIA(生物物理学的相互作用解析) > 相互作用解析の王道 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
細胞表面受容体の弱く速い認識を解析する齊藤貴士1 黒木喜美子2 福原秀雄2 前仲勝実1, 2 はじめに細胞表面の受容体とそのリガンドとなるタンパク質や糖の相互作用は、細胞の活性を制御する最初のステップであり、生体の恒常性維もにおいて重要な役割を担っています。この細胞表面受容体群によるリガンドの認識では、受容体はそれぞれのリガンドに対し適切な特異性と親和性をもっています。そして一度このバランスが崩れると、疾患の発症につながるケースが多数存在します。さらに細胞表面受容体は外界との接点であることから、微生物やウイルス由来の分子に受容体として利用されることもあります。細胞表面受容体とリガンドとの相互作用について、構造生物学的手法などから得られる情報を利用し、細胞表面受容体をターゲットとする低分子化合物あるいは抗体をはじめとするタンパク質医薬品を開発できれば、特異性の高い疾患制御が可能な薬剤開発に結びつく可能性を秘めています。
細胞表面受容体のタンパク質調製はじめに、細胞表面受容体を解析するには多くの場合、リガンドと直接結合する細胞外ドメインを抜き出して(膜貫通部位を削除して)、可溶型として発現させます。細胞表面に発現するタンパク質の多くは糖鎖修飾等の翻訳後修飾を受け、ジスルフィド結合を形成します。このため、もっとも広く用いられている大腸菌発現系では、翻訳後修飾が行われないことや、細胞内が還元状態であるためジスルフィド結合が形成されないことなどの理由で、機能的なタンパク質として大量発現できないケースが多くあります。我々はこれらを解消するために、糖鎖修飾が必須でない場合、大腸菌を用いて封入体の状態で発現させた後、正しくジスルフィド結合を形成した機能的な立体構造をとるように、巻き戻し系の確立を進めています。他方、糖鎖修飾を積極的に行うヒト培養細胞あるいは昆虫細胞を用いた発現系も構築しています。少し話がずれますが、X線結晶構造解析に適する結晶を得るために、N―グリコシルトランスフェラーゼ遺伝子を改変したヒトHEK293GnTI-細胞を用いることにより、糖鎖修飾の不均一性を抑え、均一なオリゴマンノース修飾組換えタンパク質を調製することが可能です。我々は上述の巻き戻し系とヒトHEK293GnTI-細胞発現系を基本的な発現系と位置づけてタンパク質調製を行っています(さらにカイコ個体を用いた発現系も利用するケースがあります)(図1)。
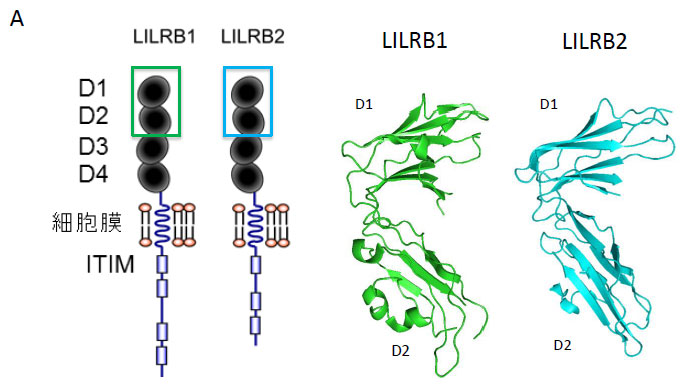
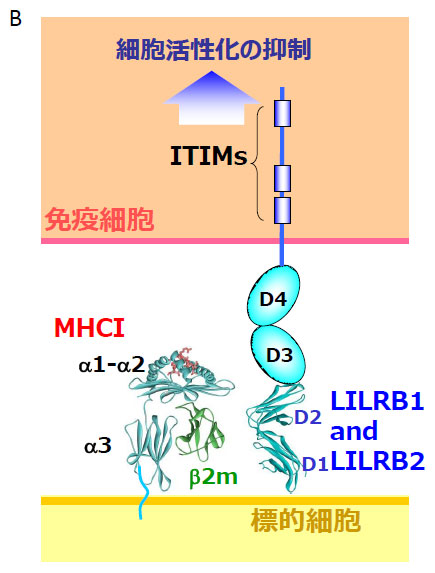
相互作用解析の実例ーLILRファミリーLILR(leukocyte immunoglobulin (Ig)-like receptor)ファミリーは1997年以降、相次いで同定されたヒト免疫細胞表面受容体群で、11個の受容体が存在します。細胞外ドメインは10 kDa程度のIg様ドメインがタンデムに2個もしくは4個連なった構造をとります。今回、とり上げるLILRB1とLILRB2は、細胞外に4つのIg様ドメインをもち、膜遠位の2つのドメイン(D1D2)を用いてリガンドMajor histocompatibility complexクラスI (MHCI、主要組織適合性抗原) を広く認識することによって、免疫細胞の抑制性シグナル伝達に関与します(図2)。
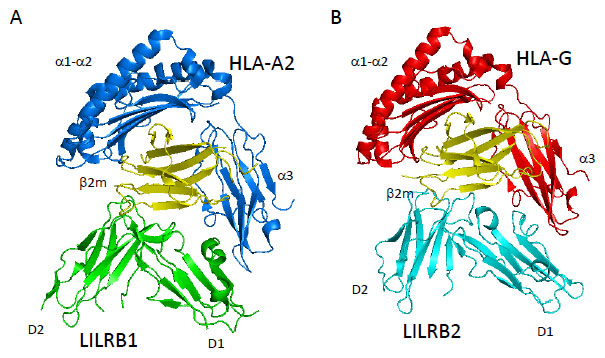
一方、リガンドとなるMHCI分子は、ヒトではHuman leukocyte antigen(HLA、ヒト白血球抗原)クラスIとよばれます。その構造は細胞外領域が3個のドメイン(α1, α2,α3)で形成される分子量約30 kDaの重鎖と、12 kDaのβ2ミクログロブリン(β2m)軽鎖、そして8~10残基程度のペプチドで形成されるヘテロ三量体です*1(図3A、B)。 *1 重鎖のα1, α2ドメインが形成するαヘリックスの溝に自己もしくはウイルス由来のペプチドが入り、これをCD8陽性T細胞に提示することで、自己・非自己の認識に関与しています。このα1, α2ドメインの著しい多型性により、MHCIは莫大な数の自己・非自己ペプチドを提示できます。
以下にLILRB1とLILRB2、そしてMHCIの相互作用に焦点をあて、X線結晶構造解析と表面プラズモン共鳴(SPR)、そして等温滴定型カロリメトリー(ITC)による解析を組み合わせた研究例について報告します。
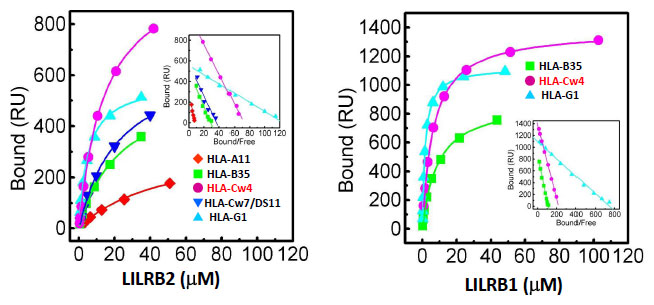
それぞれのMHCIの固定化量は 800 - 3,000 RU。 25℃で測定。
この結果、LILRB1, LILRB2ともにMHCIに対し解離定数(KD)が10-5 ~ 10-6 Mのオーダーであり、一般的な免疫細胞表面受容体の親和性と同程度であることがわかりました(表1)。また、LILRB1の方が、全体的に親和性が高いことが明らかになりました。この解析では、結合および解離速度がBiacore™の時分解能(0.2 sec)の限界に近く、測定にはhigh resolution modeでかつ1レーンずつ測定する必要があります。さらに、結合が弱いために、LILR分子を高濃度で流さなければならず、適切な測定を妨げるアグリゲーションに注意しなければなりません。測定直前に遠心して上清のみを使用することでは不十分な場合もあり、その場合は、測定直前にゲルろ過クロマトグラフィーを行い、目的分子量にあたるフラクションをそのまま用いて測定することでアグリゲーションを回避できることもあります。
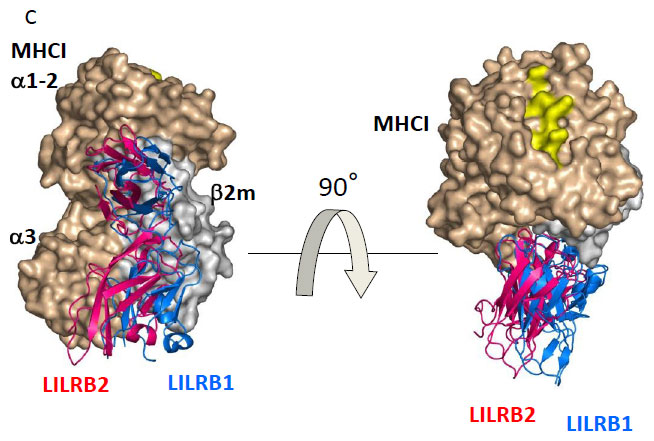
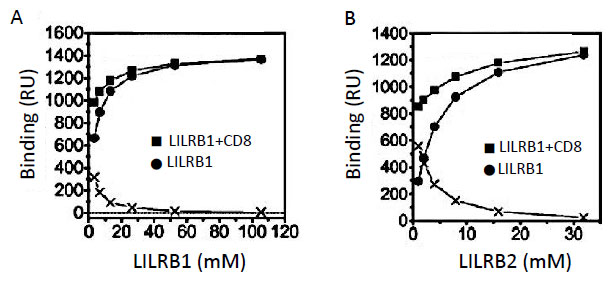
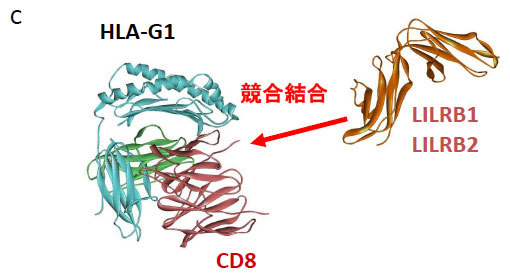
これは、HLA-G上でCD8に対しLILRB1とLILRB2の結合が競合していることを意味しています。そして、速度論的解析ではLILRB1/MHCI相互作用は免疫系受容体の中でもとりわけ早い結合、解離速度をもつことがわかりました (ka = 5.0 ~ 9.2 × 105 M-1 s-1, kd = 2.1 ~5.0 s-1)[3](表2)。
この値はMHCI分子上の結合領域が競合するCD8 (ka = 1.0 × 105 M-1 s-1, kd > 18 s-1)よりも結合速度が速く、解離速度が遅いことを示します。これらの結果から、LILRBはCD8がMHCIに結合するのを阻害することで、T細胞活性化シグナル伝達の始動を制御する、すなわちT細胞が活性化される際の閾値を上げる役割ももつことが示唆されました。このようにかなり弱い結合同士の競合実験も行うことができる点は重要であり、また速度論的な側面を比較することで生理的な意義に迫ることができる点もBiacore™を用いて解析するメリットと言えます。 さらに、LILRB1とMHCIの相互作用についてBiacore™とITC を用いた熱力学的解析を行いました[3]。Biacore™を用いた解析では、各温度に対するKDのプロットから、熱容量ΔCpの項を考慮した非線形van't Hoffの式を用いてフィッティングを行いました。一方、ITCを用いた解析はセル中のMHCIに対しLILRB1を滴定することで測定しました。その結果、両手法で決定した熱力学的パラメーターはほぼ一致するものであり、大きな差異は見られませんでした(表2)。 LILRB1によるMHCIの認識はエントロピー駆動型の相互作用であり、比熱の変化量が小さい (-0.10 ~ -0.22 kcal mol-1 K-1)ことから、結合時に大きな構造変化は起きていないと予想されました。これまでT細胞受容体やKiller cell Ig-like receptorなどの細胞表面受容体に関してリガンドとの相互作用解析が行われてきましたが、完全なエントロピー駆動型の相互作用はLILRB1が初めてでした(表3)。 ここで、エントロピー駆動型の認識とはどのようなものかを立体構造の側面から検証します。LILRB1のリガンド認識に関与するD1D2の立体構造は複数の状態で構造決定されています[3], [4], [5]。 その結果、2つの逆平行βシートで形成されるD1とD2が疎水性相互作用により鋭角に連なった構造をとりますが(図2)、LILRB1のドメイン間には可動性が存在することが明らかとなりました。また、NMRを用いてLILRB1のMHCI結合に伴う構造変化を検証すると、やはりドメイン間の可動性を示唆する結果を得ました[3]。 上述のようなLILRB1の熱力学的なパラメーター(エントロピー駆動型でかつ小さな負の熱容量)は、結合による構造変化があまりないことを示しますが、ドメイン間の可動性と矛盾します。他方、エントロピー駆動に寄与するLILRB1-MHCI間の疎水性相互作用は一般的な結合に比べ、決して多いものでなく、これで説明することもできません。そこで、我々は複合体形成後もLILRB1は十分にD1D2ドメイン間の可動性が残り、揺らいだ認識モードが存在すると考えています。これは速度論的に速い認識をも説明できるモデルです。 このように熱力学的側面を調べることで、その分子認識がより明らかになります。上述のような比較的弱い結合(サブμMオーダー以下)は、サンプル量が多く必要である点や高濃度にしなければいけない点など解析には難しい点があり、まだ十分なデータが蓄積されておらず、今後のデータベースの充実が期待されます。また、van't Hoffのモデルにより決定したパラメーターと直接熱量を測定したパラメーターが一致しないことを指摘する論文がありますが、我々の研究対象である比較的弱い結合で、かつ速度論的に速い認識に関しては、かなり一致するようです。この点もデータの蓄積によりその本質が明らかとなってくることが期待されます。
ΔG:ギブスエネルギー変化、 ΔH:エンタルピー変化, -TΔS:エントロピー変化, ΔCp:比熱変化
References
「相互作用解析の王道」について「相互作用解析の王道」は、2009年8月よりバイオダイレクトメールでお届けしています。
バイオダイレクトメールは弊社WEB会員向けメールマガジンです。バイオダイレクトメールの配信をご希望の方は、下記リンク先からご登録をお願いいたします。 関連リンク津本先生の研究内容や論文などはこちら→津本浩平先生の研究室Webサイト お問合せフォーム※日本ポールの他事業部取扱い製品(例: 食品・飲料、半導体、化学/石油/ガス )はこちらより各事業部へお問い合わせください。 お問い合わせありがとうございます。 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
© 2026 Cytiva